Die Intensiv-Antwort
Ich habe ja immer noch nicht so recht meinen Frieden gemacht mit dem Schluss von 66 ist das neue 50 vom letzten Freitag, dass nämlich die Impfkampagne bisher fast keinen Unterschied macht für unsere Fähigkeit, hohe Inzidenzen – die wir praktisch sicher bald haben werden, wenn wir nicht wieder viel weiter zumachen als derzeit – zu ertragen, ohne dass die Intensivstationen zu einem Schlachtfeld werden.
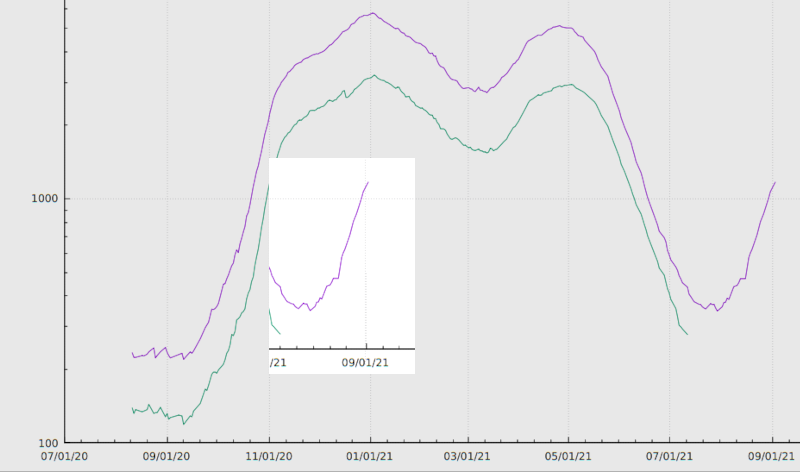
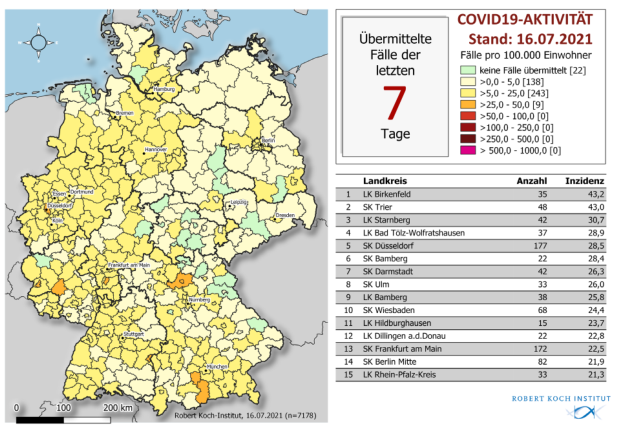
Die Frage lässt sich zurückführen auf: „Wie viele Intensivbetten waren eigentlich relativ zur jeweiligen Inzidenz durch SARS-2-Erkrankte belegt?“ (bei allen berechtigten Zweifeln an der zuverlässigen Messung letzterer). Diese Antwort der Intensivbelegung auf das Infektionsgeschehen – ich taufe es mal int/inc, weil ich zu faul bin, nachzusehen, wie die Profis das nennen – kriege ich mit dem Besteck vom Coronafilm-Post und meiner DIVI-Auswertung relativ schnell ausgerechnet, und wenn ich dann das Verhältnis von DIVI-Belegung (gezogen jeweils aus den RKI-Berichten der Zeit) zur bundesweiten Inzidenz (auf der Basis der Referenz-Daten des RKI) plotte, sieht das so aus:
Ich habe die y-Achse wieder logarithmisch eingeteilt, in dem Fall nicht, weil ich irgendwelche exponentiellen Verläufe erwarten würde, sondern weil mir im Wesentlichen der Verlauf im unteren Teil des Wertebereichs am Herzen liegt.
Das dominierende Feature Anfang Juli 2021 nämlich ist schnell erklärt: Hier waren die Inzidenzen sehr niedrig, die langwierigen Fälle blieben aber auf den Intensivstationen. Und so ging es bis zu 120 IntensivpatientInnen pro Punkt Inzidenz. Ähnlich reflektiert das zweithöchste Feature rund um den Februar 2021 lediglich die fallende Inzidenz und also quasi vorauseilend kleine Nenner zwischen zweiter und dritter Welle.
Wenn mensch sich diese großen Features mal rausdenkt, kommt als Basis etwas heraus zwischen 20 und 40 Intensivpatient_innen pro Inzidenzpunkt während der Wellen. Mitte August, als die Inzidenzen kräftig anzogen, haben wir vielleicht auch mal 10 touchiert, was zu erwarten wäre, wenn die Basisrate 30 wäre und um die zwei Drittel der Leute geimpft sind. Angesichts der dramatischen Altersabhängigkeit der Hospialisierung von SARS-2 sind solche Überlegungen aber ohne demographische Betrachtungen unsinnig.
Der Anstieg ganz am rechten Rand des Graphen dürfte übrigens zumindest in diesem Ausmaß ein Artefakt sein, denn ich rechne hier wie gesagt mit dem, was das RKI als Referenzdatum angibt (wo das Infektionsdatum das Meldedatum ersetzt, wenn das rauszukriegen ist), und es kann so sein, dass aus den letzten zwei Wochen Fälle in die weitere Vergangenheit gewandert sind, die, die sie in Zukunft aus der Gegenwart bekommen werden, aber noch nicht da sind; das würde die Inzidenz unterschätzen und mein int/inc überschätzen.
Nehmen wir trotzdem an, wir hätten mit der derzeitigen Virenpopulation, Infektionsdemographie und Impfquote im Gleichgewicht 20 Intensivpatient_innen pro Indizenzpunkt. Wenn wir dann unter 6000 Intensivpatient_innen bleiben wollen, ist eine 300er-Inzidenz das äußerste, was mit viel Glück zu stemmen wäre. Wenn also nicht mein int/inc drastisch runtergeht – als Faustregel dürfte taugen, dass jede Halbierung des Anteils der ungeimpften Erwachsenen auch eine Halbierung von int/inc nach sich ziehen würde –, wird es noch lang dauern, bis die (relativ) strikte Kontrolle von SARS 2 aufhören kann.
Eine 300er-Inzidenz in der RKI-Rechnung heißt ja, dass jede Woche 300 von 100000 Menschen SARS-2 hinter sich bringen. Bis dann alle mal SARS-2 hatten (und alle werden es irgendwann gehabt haben, dauerhaft sterile Impfungen gibts bei Atemwegserkrankungen nicht), dauert es logischerweise 100000/300, also gut 300 Wochen. Oder grob sechs Jahre.
Sorry, liebe Ungeimpfte: so lange will ich echt nicht warten, bis ich nicht mehr an jeder Ecke damit rechnen muss, Namen und Geburtsdatum (im Impfzertifkat) von mir nicht annähernd kontrollierbaren Rechnern überantworten zu müssen.
Technics
Der Code, der das erzeugt, ist nicht spannend; mit corona.py sieht die Inzidenzberechnung so aus:
def get_incidences(tuples):
"""returns a mapping of dates to 7-day incidences from RKI tuples
as yielded by corona.iter_counts.
"""
tuples.sort(key=lambda r: r[0])
incidences = {}
queue = collections.deque([0], maxlen=7)
cur_date = None
for tup in tuples:
if tup[0]!=cur_date:
if len(queue)==7:
incidences[cur_date] = sum(queue)/830
queue.append(0)
cur_date = tup[0]
queue[-1] += tup[2]
incidences[cur_date] = sum(queue)/83e4
return incidences
with open(corona.DATA_DIR
+"Aktuell_Deutschland_SarsCov2_Infektionen.csv") as f:
all_rows = list(corona.iter_counts(f, True))
incidences = get_incidences(all_rows)
Das Zusammenfummeln mit den DIVI-Daten nimmt meine aus den RKI-Berichten gescreenscrapten Daten. Das ist an sich Quatsch, und ihr solltet die Daten einfach von der DIVI selbst ziehen, wenn ihr das reproduzieren wollt.
Mit dem zusammengefummelten Daten (Spalten: Datum, int/inc, Intensivbelegung und referenzdatenbasierte Inzidenz) könnt ihr aber selbst Plots machen. Das Bild oben kommt aus diesem Gnuplot-Skript:
set size ratio 0.375 set key inside left top set xdata time set yrange [5:200] set ytics (10,22,47,100) set timefmt "%Y-%m-%d" set xrange ['2020-08-01':'2021-08-31'] set logscale y set xtics format "%Y-%m-%d" time set term svg size 800,300 set out "int-vs-inc.svg" plot "int-vs-inc.txt" using 1:2 with lines title "int/inc"
ElektronikbastlerInnen werden die Ticks auf der y-Achse wiedererkennen. Logarithmen sind überall.
![[RSS]](../theme/image/rss.png)