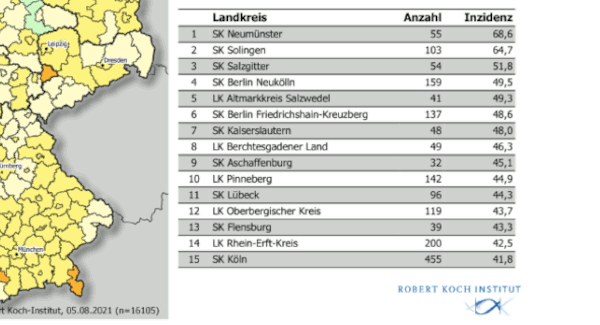
Anlass meiner Paranoia: Im RKI-Bericht von heute drängeln sich verdächtig viele Kreise gerade unter der 50er-Inzidenz.
Mein Abgesang auf die RKI-Berichte von neulich war wie erwartet etwas voreilig: Immer noch studiere ich werktäglich das Corona-Bulletin des RKI. Es passiert ja auch wieder viel in letzter Zeit. Recht schnell schossen die ersten Landkreise im Juli über die 50er-Schwelle, während die breite Mehrheit der Kreise noch weit von ihr entfernt war. Das ist, klar, auch so zu erwarten, wenn die „Überdispersion“ (find ich ja ein komisches Wort für „die Verteilung der Zahl der von einem_r Infizierten Angesteckten hat einen langen Schwanz nach oben hin“, aber na ja) noch irgendwie so ist wie vor einem Jahr, als, wie im inzwischen klassischen Science-Artikel von Laxminarayan et al (DOI 10.1126/science.abd7672) auf der Grundlage von Daten aus Indien berichtet wurde, 5% der Infizierten 80% der Ansteckungen verursachten (und umgekehrt 80% der Infizierten gar niemanden ansteckten): SARS-2 verbreitete sich zumindest in den Prä-Alpha- und -Delta-Zeiten in Ausbrüchen.
Nachdem aber die ersten Landkreisen die 50 gerissen hatten, tat sich für eine ganze Weile im Bereich hoher Inzidenzen nicht viel; auch heute sind nur drei Landkreise über der 50er-Inzidenz, während sich knapp darunter doch ziemlich viele zu drängen scheinen.
Und da hat sich ein Verdacht in mir gerührt: Was, wenn die Gesundheitsämter sich mit Händen und Füßen wehren würden, über die vielerorts immer noch „maßnahmenbewehrte“ 50er-Schwelle zu gehen und ihre Meldepraktiken dazu ein wenig… optimieren würden? Wäre das so, würde mensch in einem Histogramm der Inzidenzen (ein Häufigkeit-von-Frequenzen-Diagram; ich kann die nicht erwähnen ohne einen Hinweis auf Zipfs Gesetz) eine recht deutliche Stufe bei der 50 erwarten.
Gibt es die? Nun, das war meine Gelegenheit, endlich mal mit den Meldedaten zu spielen, die das RKI bereitstellt – zwar leider auf github statt auf eigenen Servern, so dass ich mit meinen Daten statt mit meinen Steuern bezahle (letzteres wäre mir deutlich lieber), aber das ist Jammern auf hohem Niveau. Lasst euch übrigens nicht einfallen, das ganze Repo zu klonen: Das sind ausgecheckt wegen eines gigantischen Archivs krasse 24 GB, und was ihr tatsächlich braucht, sind nur die aktuellen Zahlen (Vorsicht: das sind auch schon rund 100 MB, weil das quasi die ganze deutsche Coronageschichte ist) und der Landkreisschlüssel (vgl. zu dem Update unten).
Auch mit diesen Dateien muss mensch erstmal verstehen, wie aus deren Zeilen die Inzidenzen werden, denn es ist nicht etwa so, dass jede Zeile einer Erkrankung entspricht: Nein, manche berichten mehrere Fälle, es wird nach schon gemeldeten und ganz neuen Fällen unterschieden, und eventuell gibts auch noch Korrekturzeilen. Dazu findet ein in-band-signalling zu Gestorbenen und Genesenen statt. Lest das README aufmerksam, sonst verschwendet ihr nur (wie ich) eure Zeit: EpidemiologInnen denken ganz offenbar etwas anders als AstronomInnen.
Das Ergebnis ist jedenfalls das hier:
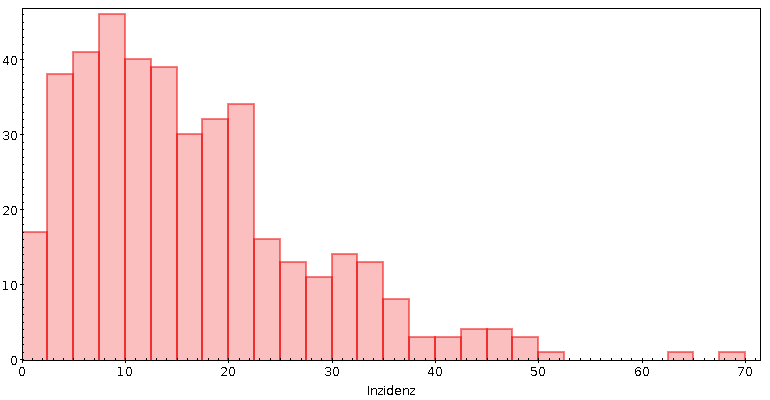
Ich muss also meinen Hut essen: Wenn da irgendwo Hemmschwellen sein sollten, dann eher knapp unter 40, und das ist, soweit ich weiß, in keiner Corona-Verordnung relevant. Na ja, und der scharfe Abfall knapp unter 25 könnte zu denken geben. Aber warum würde jemand bei der 25 das Datenfrisieren anfangen? Der Farbe im RKI-Bericht wegen? Nee, glaub ich erstmal nicht.
Wenn ihr selbst mit den RKI-Daten spielen wollt, kann euch das Folgende vielleicht etwas Fummeln ersparen – hier ist nämlich mein Aggregationsprogramm. Ich werdet die Dateipfade anpassen müssen, aber dann könnt ihr damit eure eigenen Inzidenzen ausrechnen, ggf. auch nach Altersgruppen, Geschlechtern und was immer. In dem großen CSV des RKI liegt in der Tat auch die Heatmap, die jetzt immer im Donnerstagsbericht ist. Reizvoll fände ich auch, das gelegentlich zu verfilmen…
Hier jedenfalls der Code (keine Abhängigkeiten außer einem nicht-antiken Python). So, wie das geschrieben ist, bekommt ihr eine Datei siebentage.csv mit Landkreisnamen vs. Inzidenzen; die entsprechen zwar nicht genau dem, was im RKI-Bericht steht, die Abweichungen sind aber konsistent mit dem, was mensch von lebenden Daten erwartet:
# (RKI-Daten zu aktuellen 7-Tage-Meldeinzidenzen: Verteilt unter CC-0)
import csv
import datetime
import sys
LKR_SRC = "/media/incoming/2020-06-30_Deutschland_Landkreise_GeoDemo.csv"
INF_SRC = "/media/incoming/Aktuell_Deutschland_SarsCov2_Infektionen.csv"
LANDKREIS = 0
MELDEDATUM = 3
REFDATUM = 4
NEUER_FALL = 6
ANZAHL_FALL = 9
def getcounts(f, n_days=7):
counts = {}
collect_start = (datetime.date.today()-datetime.timedelta(days=n_days)
).isoformat()
sys.stderr.write(f"Collecting from {collect_start} on.\n")
row_iter = csv.reader(f)
# skip the header
next(row_iter)
for row in row_iter:
if row[MELDEDATUM]>=collect_start:
key = int(row[LANDKREIS])
kind = row[NEUER_FALL]
if kind!="-1":
counts[key] = counts.get(key, 0)+int(row[ANZAHL_FALL])
return counts
def get_lkr_meta():
lkr_meta = {}
with open(LKR_SRC, "r", encoding="utf-8") as f:
for row in csv.DictReader(f):
row["IdLandkreis"] = int(row["IdLandkreis"])
row["EW_insgesamt"] = float(row["EW_insgesamt"])
lkr_meta[row["IdLandkreis"]] = row
return lkr_meta
def main():
lkr_meta = get_lkr_meta()
with open(INF_SRC, "r", encoding="utf-8") as f:
counts = getcounts(f)
with open("siebentage.csv", "w", encoding="utf-8") as f:
f.write("Lkr, Inzidenz\n")
w = csv.writer(f)
for lkr in lkr_meta.values():
w.writerow([lkr["Gemeindename"],
1e5*counts.get(lkr["IdLandkreis"], 0)/lkr["EW_insgesamt"]])
if __name__=="__main__":
main()
Nachtrag (2021-08-13)
Eine Woche später ist der Damm definitiv gebrochen. Von drei Landkreisen über dem 50er-Limit sind wir laut aktuellem RKI-Bericht jetzt bei 39, wobei allein seit gestern 10 dazukamen. An die aktuelle Verteilung würde ich gerne mal eine Lognormalverteilung fitten:
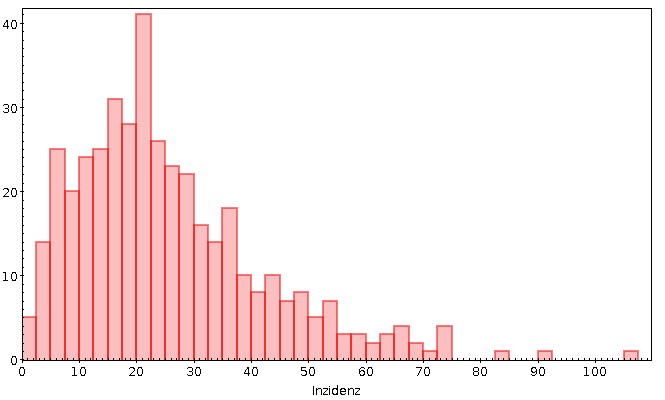
Nicht, dass ich eine gute Interpretation hätte, wenn das lognormal wäre. Aber trotzdem.
Nachtrag (2021-09-08)
Das RKI hat die Landkreis-Daten (2020-06-30_Deutschland_Landkreise_GeoDemo.csv) aus ihrem Github-Repo entfernt (commit cc99981f, „da diese nicht mehr aktuell sind“; der letzte commit, der sie noch hat, ist 0bd2cc53). Die aus der History rausklauben würde verlangen, das ganze Riesending zu clonen, und das wollt ihr nicht. Deshalb verteile ich unter dem Link oben die Datei unter CC-BY 4.0 International, mit Namensnennung… nun, Destatis wahrscheinlich, oder halt RKI; die Lizenzerklärung auf dem Commit ist nicht ganz eindeutig. Als Quelle der Geodaten war vor der Löschung https://www.destatis.de/DE/Themen/Laender-Regionen/Regionales/Gemeindeverzeichnis/Administrativ/Archiv/ angegeben, aber da finde ich das nicht.
Zitiert in: Corona als Film
![[RSS]](./theme/image/rss.png)